はじめに
RSNA Intracranial Aneurysm Detection という Kaggle コンペに参加しました。自分の復習も兼ねて上位(1st〜7th)の解法を読み、その内容をまとめます。特に各解法に出てくる図を理解したかったので、図をメインに整理しています。
筆者がポンコツなので、記事にミスがある・重要なところを見逃している可能性があります。記事の最後に各解法へのリンクをつけていますので、必ず原典も参考にしてください。(もし間違いに気づいたら、こっそり教えてもらえると助かります)
コンペ概要
説明
-
頭蓋内脳動脈瘤は世界人口の約 3%に影響を与え、毎年 50 万人以上が亡くなっており、その約半数は 50 歳未満です。
-
今回のコンペでは、様々な施設から得られたデータを用いて、実際の臨床で起こるようなバイアス(施設ごとの撮像条件の差や、画像コントラストの違いなど)を考慮しつつ、汎用的に動くモデルの構築が課題とされました。
-
評価指標は、動脈瘤が存在するかどうか(aneurysm_present; AP)と、脳動脈瘤の位置に対応した 13 個のラベルを合わせた 14 ラベルに対する AUC です。
データ
学習には CTA、MRA、T1 MRI、T2 MRI など、複数モダリティで撮影された約 4,000 症例分の画像が提供されました。
-
seriesは各患者ごとに 1 セット与えられ、その中に頭部全体の 3D 画像がスライスとして並べられています。データ形式は DICOM です。 -
train.csvには動脈瘤の有無と 13 個の位置ラベルのほか、年齢や性別などの情報が含まれています。 -
train_localizer.csvでは、動脈瘤の中心座標が与えられています。 -
一部の series(約 200 個)には、血管セグメンテーションも提供されていました。
上位解法の全体的な傾向
- どのチームも何らかの マルチステージ構成(ROI 抽出 → 本タスク) を採用していました。
- 3D モデル(nnU-Net, 3D ResNet など)を軸にしつつ、2D ベースのモデルや外部データも積極的に活用していました。
1 位解法
1st stage : nnU-Net を用いた ROI 抽出

Preprocessed volume
- DICOM データを NIfTI に変換し、その後座標系を RAS に揃えたうえで読み込み、nnU-Net の標準前処理を適用しています。
Coarse Scan
-
脳スキャンのボリュームを等方 1 mm にリサンプリングし、粗い nnU-Net(Model1)に入力します。このモデルは各ボクセルに対して 13+1 クラスではなく、4 クラス(0: 背景 + 3 つの血管グループ)のラベルを出力します。
-
後処理としては、
- 出力ラベルを「血管あり / 血管なし」の 2 値に変換
- 血管ありボクセルの座標をすべて取得
- それらに対して
DBSCANによるクラスタリングを実施 - 最も大きいクラスタの重心座標を計算
- その座標を中心として 140 mm の立方体 ROI をクロップし、ボクセルとして切り出す
…という流れになっています。
Fine Inference
-
先ほど抽出した ROI を
(0.80, 0.45, 0.44)のボクセル間隔にリサンプリングします(この値は nnU-Net の学習結果から採用したと書かれていました)。この ROI を、設定の異なる 2 つの nnU-Net(Model2 / Model3)に入力します。 -
Model2 の損失関数は
Dice + CE + SkeletonRecall(weight=1)、Model3 はTversky + CE + SkeletonRecall(weight=3)です。前者はバランス重視、後者は見逃しを極力減らすことを狙っていると考えられます。 -
ROI refineでは、Model2 のセグメンテーション結果から最小の bbox を作成し、そこに margin を追加して新しい ROI を定義します。それを分類モデルの入力サイズに合わせて補間し、最終的なFinal ROIを作成します。このとき同時に、13 クラスの血管マスクも Model2/Model3 から出力しています。
2nd stage : 13 クラス + 動脈瘤有無 + 補助タスク
nnU-Net Backbone
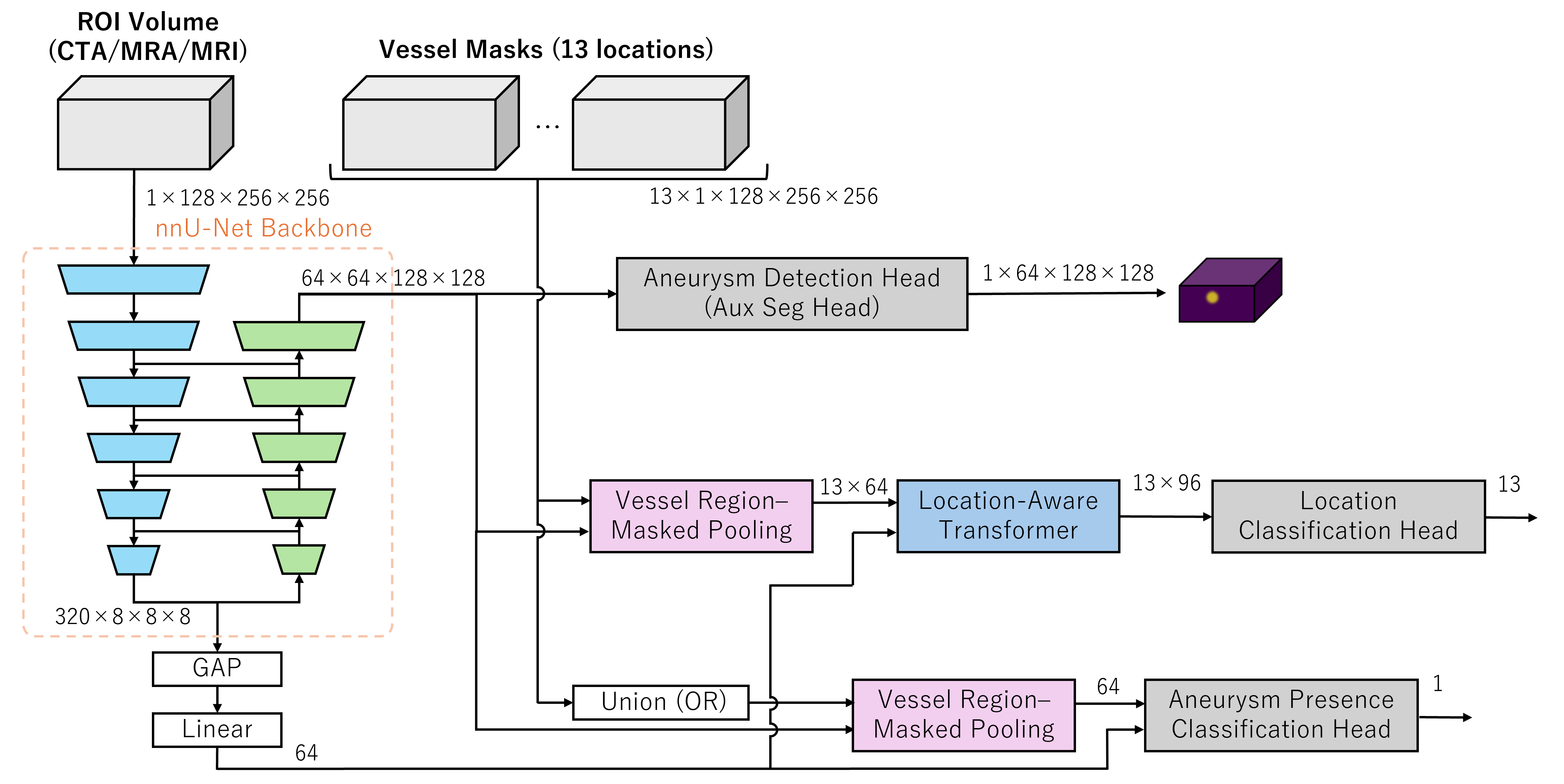
-
この図は、1st stage で抽出した情報を使って最終的な予測をどのように出しているかを示したものです。
-
左上の
ROI Volumeが先ほどのFinal ROIに対応します。Vessel maskは Model2/3 の出力を統合したものです。 -
nnU-Net Backbone では、
1 × 128 × 256 × 256の ROI から、エンコーダ最終層で320 × 8 × 8 × 8、デコーダ最終層で64 × 64 × 128 × 128の特徴マップを出力します。前者は最終的に ROI の特徴を集約した 64 次元のベクトルになり、後者は補助タスクや Union(動脈瘤が存在するかどうか)、13 クラスの推定に用いられます。 -
Aneurysm Detection Headでは、nnU-Net デコーダの特徴マップに Conv ヘッドを付け、補助タスクとして「動脈瘤の存在する球」を予測し、そのロスを計算しています。この教師データには、おそらくtrain_localizer.csvの動脈瘤座標を使っていると思われます。
Vessel Region-Masked Pooling
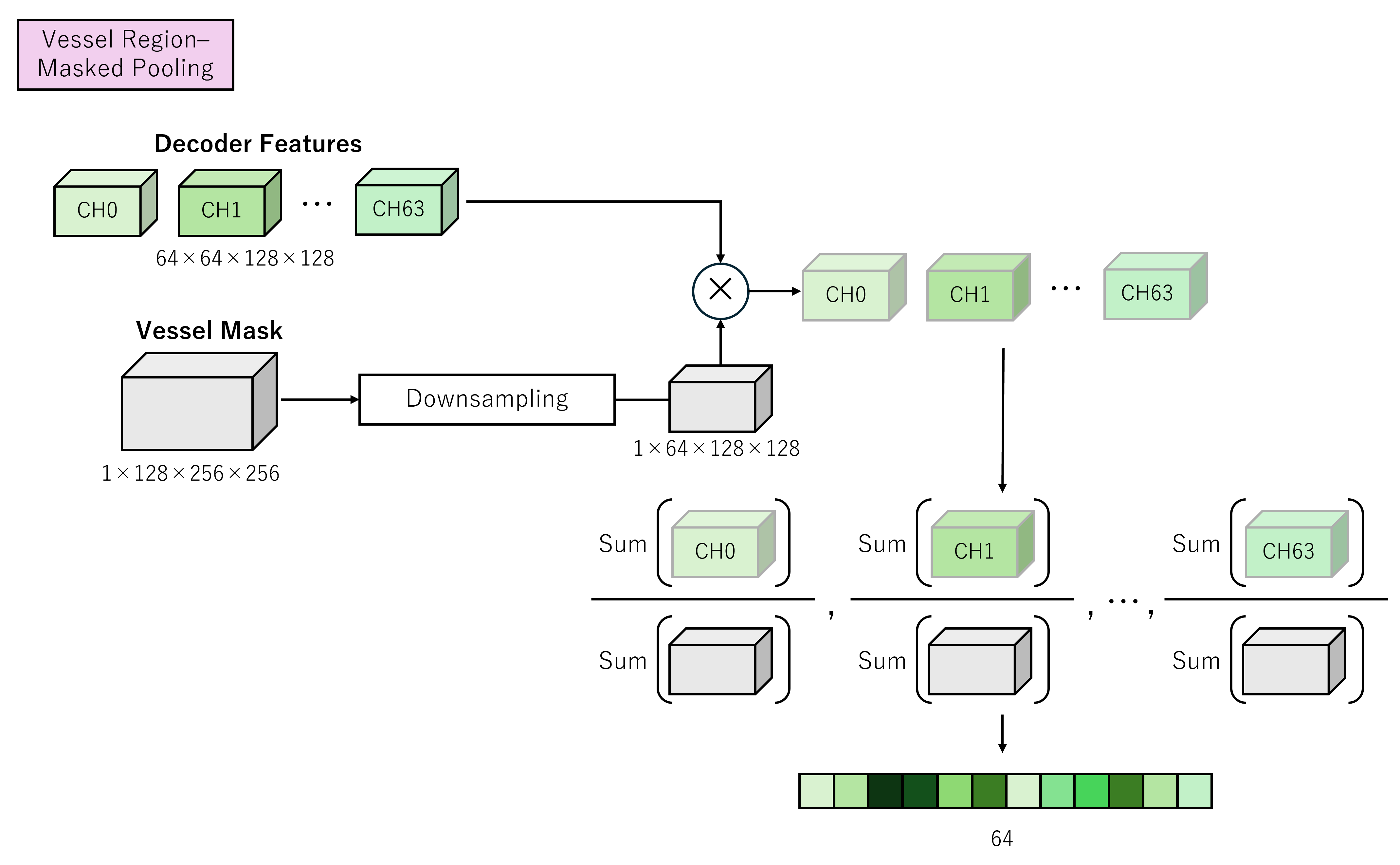
-
この図は、上の図にも出てきた
Vessel Region-Masked Poolingを説明しています。まず、Vessel Mask をデコーダ出力と同じ解像度にダウンサンプリングし、Decoder の特徴量と要素ごとに掛け合わせます。 -
Mask は 0〜1 の値を取るので、Mask が 0 の位置の特徴は 0 に、1 の位置の特徴はそのまま残ります。これにより「どこに注目すべきか」を明示した状態で特徴量を集約できるようになります。掛け合わせた特徴量をチャネルごとに、マスクが 1 の領域内で平均してベクトルとして出力します。
-
1 つ前の図では
Unionと書かれていましたが、これは 13 クラスを「動脈瘤が存在するかどうか」の 1 クラスにまとめたもので、処理自体は同様です。
Location-Aware Transformer
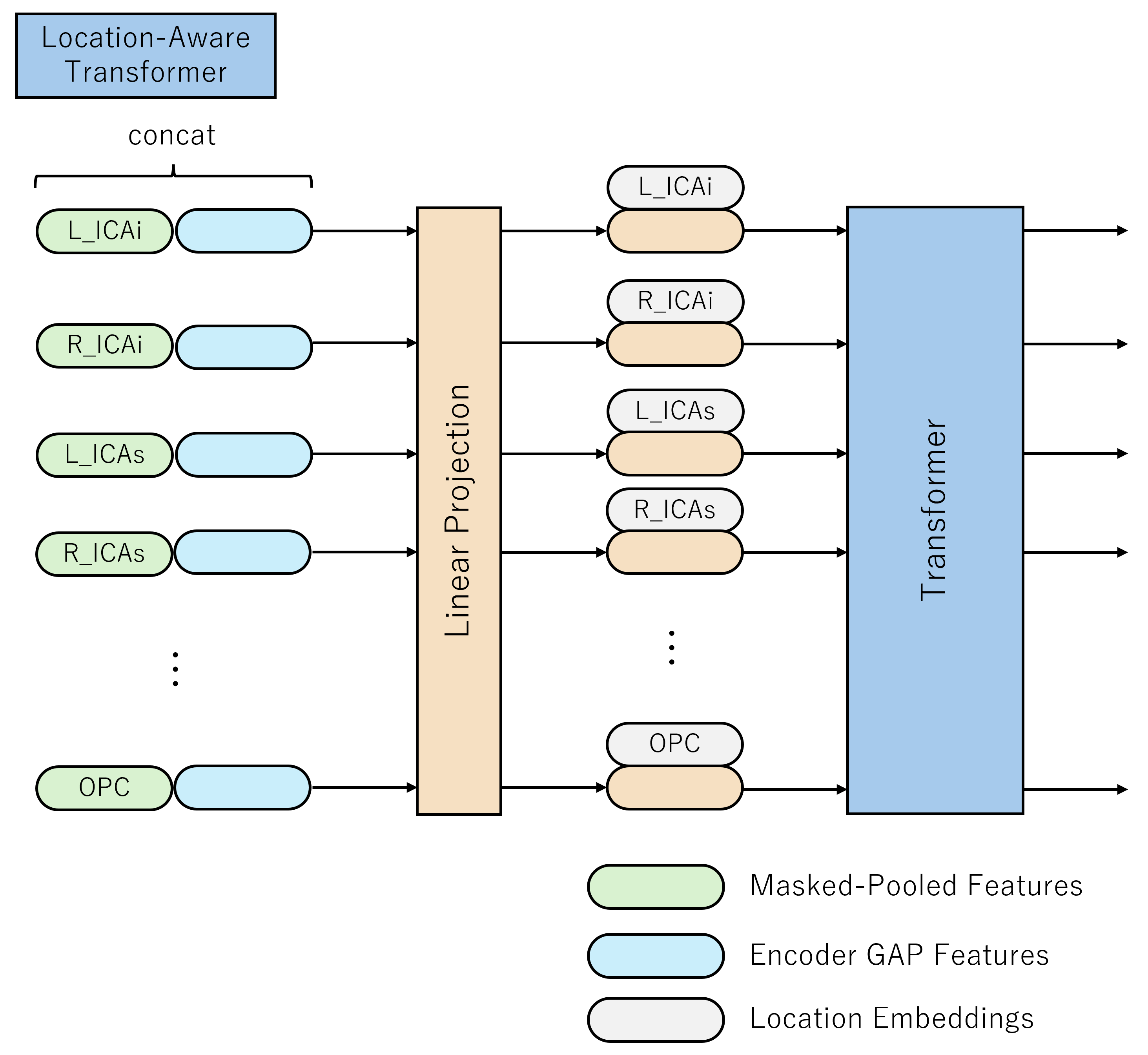
-
この図は、2 つ前の図の
Location-Aware Transformerに入力される特徴量の構成を説明しています。 -
緑色のベクトルが、先ほどの Vessel Mask によるマスク付きプーリングで得た 13 クラス分の特徴量です。これに、nnU-Net エンコーダから抽出したグローバルな特徴量を複数コピーして結合し、
Linear Projectionブロックで Transformer 用の埋め込みベクトルに射影しています。
その他の工夫
- フェイルセーフ機構を用いて、セグメンテーションや ROI 抽出で異常が発生した場合は、事前に計算しておいた OOF の確率を返すようにしていた。
- 推論時には TTA(Flip など)を使用。
- 強めの Data Augmentation を適用。
- TensorRT による推論高速化。
2 位解法
データの前処理
-
今回のデータセットは、①モダリティがバラバラ、②スライス間隔(SliceThickness / SpacingBetweenSlices)がバラバラ、③1 ファイルに複数フレームが入っているケースがある、などの理由で、画一的な前処理が難しい問題設定でした。
-
DICOM データを pydicom で読み込み、SliceThickness 等のメタデータが存在しない場合でもスライス間隔を推定できるようなロジックを用意していました。
-
また、T2 画像は他のシリーズとは画像の向きが異なることがあるため、T2 専用の方向分類器 を作成し、向きの正規化を行っていました。
1st stage
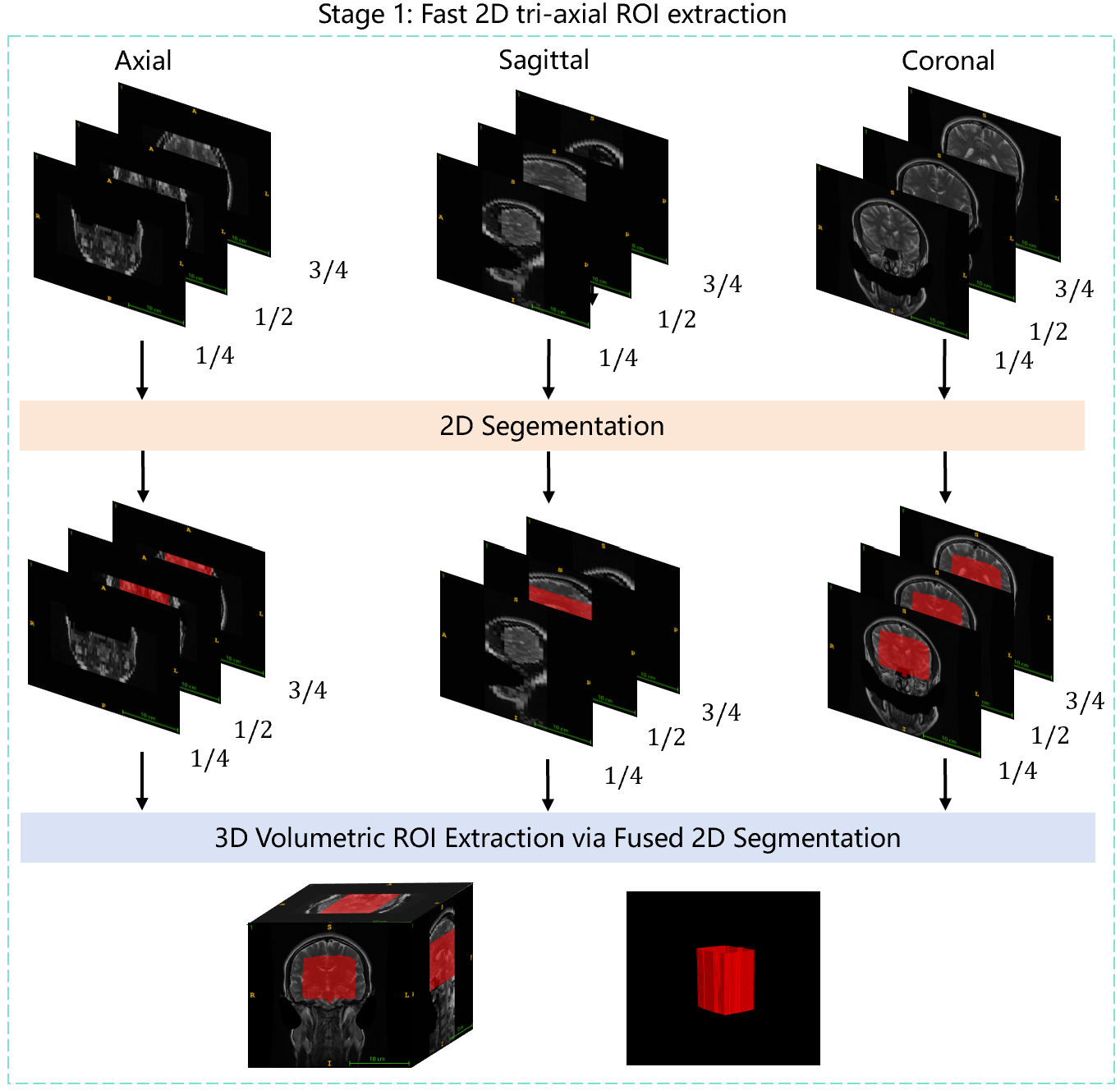
-
axial / sagittal / coronal の 3 方向それぞれから 3 枚ずつ、計 9 枚のスライスを取得します。
-
これら 2D 画像を 2D nnU-Net に入力し、背景と血管の 2 値マスクを作成します。
-
axial / sagittal / coronal それぞれの方向でマスクが得られるので、それらを組み合わせることで 3D の ROI をクロップします。
2nd stage
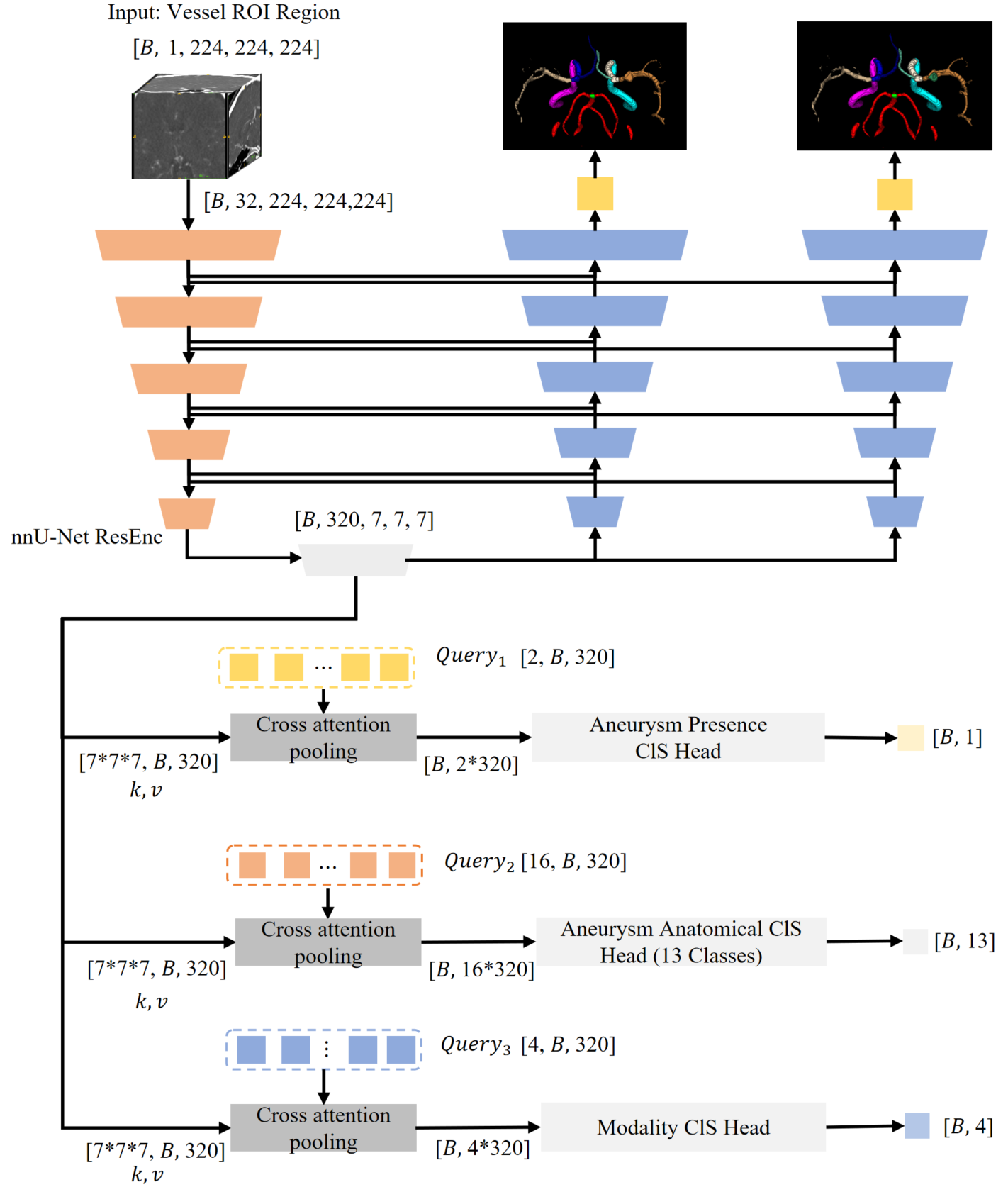
-
この図は、1st stage で作成した ROI を入力とした nnU-Net + Cross Attention による分類機構を示しています。
-
nnU-Net ResEnc からの出力は
[B, 320, 7, 7, 7]で、これを[7*7*7, B, 320]に reshape し、Cross Attention Pooling に入力します。ここで、動脈瘤の有無、13 クラス分類、モダリティ分類で query を分け、それぞれ別の Head を付けています。 -
また、血管セグメンテーション(図の左側)と動脈瘤セグメンテーション(右側)を同時に学習しており、これにより学習が安定したと報告されています。
セグメンテーションについて
-
元の RSNA データには血管セグメンテーションが一部(約 200 例)しかありませんが、両 stage でセグメンテーションを利用している点が気になったので、論文を少し深掘りしてみました。
-
結論としては、TopCoW / TopBrain という外部データセットから事前学習したセグメンテーションモデルを使い、疑似ラベルを作りながら学習を回している という形です。
1st stage のセグメンテーション
- 外部データセットには CTA/MRA の血管セグメンテーションが付いているので、これをそのまま 2D セグメンテーションタスクとして学習し、そのモデルを使って ROI を作っています。
2nd stage のセグメンテーション
-
血管セグメンテーションについては、TopCoW / TopBrain データセットに、ウィリス動脈輪などの フル 3D 血管セグメ + 解剖学的クラスラベル が付いているので、まずこれで CoW セグメントモデルを作成します。それを RSNA データに適用して疑似血管マスクを生成し、手動での微修正も加えて pseudo mask を作成しています。
-
動脈瘤セグメンテーションは、RSNA の
train_localizer.csvにある動脈瘤中心座標を用いて、半径 2〜5 mm のランダムな立方体を作成し、それをボクセルマスクに変換します。この粗いボックスマスクを教師として U-Net を学習し、その出力をさらに洗練させていきます。具体的には:- モデル出力を閾値でバイナリ化
- 3D connected component で塊に分割
- 中心点から最も近い塊だけを残す
- 血管セグメンテーションを用いて、血管からあまりに離れている領域を削除
- それを新しい教師マスクとして再学習
…というように、pseudo label を iterative に洗練しているようです。
その他の工夫
- TTA では、画像・ラベルを左右反転したものも追加していました。
- 前処理部分をかなり作り込んでおり、「読めない DICOM があってフォールバックする」のではなく、すべての画像を確実に読み込めるように 実装していたそうです。
3 位解法
1st stage
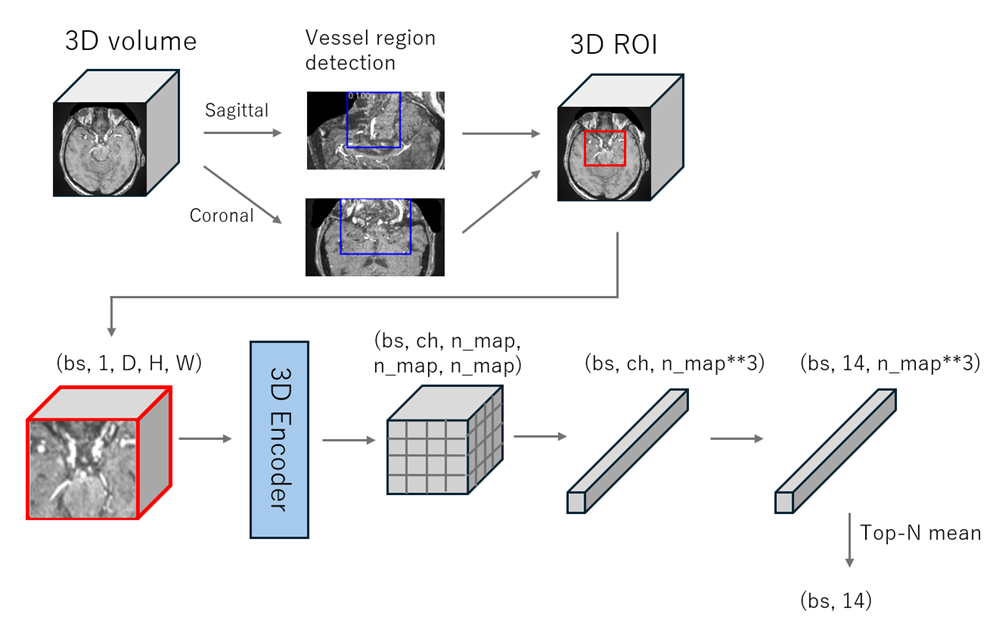
-
図の上半分が 1st stage です。
-
まず、3D volume の血管セグメンテーションマスクから、Sagittal / Coronal 方向の 2D MIP(最大値投影)画像を生成します。このセグメンテーションマスクをどう作成しているかは論文中で明確ではなく、正直よく分かりませんでした……。おそらく、与えられている一部の血管セグメンテーション(約 200 例)を教師にしているか、外部データで事前学習している可能性があります。
-
作成した 2D MIP 画像に対して YOLOv8 で BBox 検出を行い、その BBox を基に 3D の ROI を作成します。
2nd stage
-
図の下半分が 2nd stage です。
-
1st stage で得られた 3D ROI を 3D ResNet-18 に入力します。3D モデルなので出力も 3D 特徴マップですが、デフォルトでは空間解像度が
4 × 4 × 4になります。ここを25 × 25 × 25のように高解像度に変更することで、性能が向上したと報告されていました。 -
最後に、この 3D 特徴マップを全結合層にかけ、14 クラス分類を行います。特徴マップ内のセルごとに予測を出しており、背景セルが多数を占めるため、推論時には Top-N のセルだけを集約して平均を取る(Top-N mean)という工夫を入れています。
その他の工夫
- テストデータの DICOM には spacing に関するタグがなく、1 ピクセルあたりの mm 単位やスライス間隔が分かりませんでした。そこで、各症例の画像を z 軸方向に積み重ねた volume を入力として、EfficientNetV2-S で
x_spacing, y_spacing, z_spacingを回帰し、推定 spacing を使って前処理を行っていました。
4 位解法
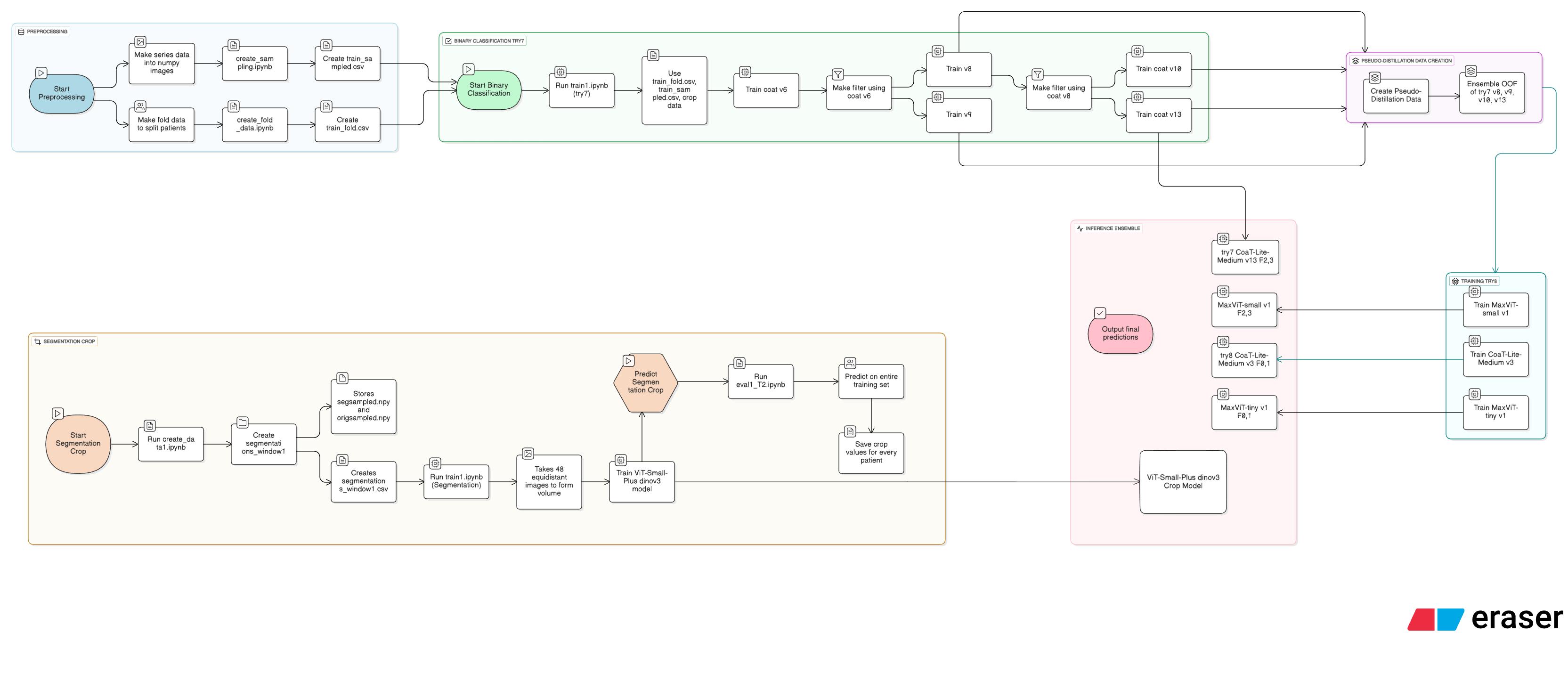
図にある 青(Preprocessing)→ オレンジ(Segmentation Crop)→ 黄緑(Binary Classification try7)→ 紫(Pseudo‑Distillation)→ 水色(Training try8)→ ピンク(Inference Ensemble) の流れに沿って説明します。
Preprocessing(青)
-
train.csvを読み込み、各シリーズごとにサイズが揃っているスライスのみを採用します。例えば CT の試し撮りのようなスライスは画像サイズが異なることが多いため、それらを除外するイメージです。 -
各スライスを
.npyとして保存し、同時にスライス単位のメタ情報テーブルtrain_sampled.csvも作成します。 -
modality をラベルにして stratified K-fold を行い、その fold 情報を
train_fold.csvとして保存します。
Segmentation crop(オレンジ)
-
各患者の DICOM から 3D volume を構築し、軸方向に等間隔で 48 枚をサンプリングして
.npy(volume)に保存します。 -
さらに、元の動脈瘤セグメンテーションマスクから画像内での bbox(
x1, x2, y1, y2)を計算し(座標は 0〜1 に正規化)、それらを 1 行にまとめたsegmentations_window1.csvを作成します。 - ここで ViT-Small-Plus DINOv3 を使った bbox 回帰モデルを学習します。
- 入力:
segmentations_window1.csvのwindow_fileから読み込んだ volume を、48 枚スライス → 3ch × 16 枚画像に reshape し、最終的に[batch, 16, 3, 128, 128]形式に変換。 - 出力: 正規化済み bbox 座標(
x1, x2, y1, y2)。 - 損失: L1(MAE)。4-fold で学習。
- 入力:
- 学習した Crop モデルを使って、全 train/test の volume に対して bbox を推論し、患者ごとの ROI を取得します。
Binary classification try7(黄緑)
-
各スライス画像を患者ごとの crop bbox で切り抜き、
aneurysm_present + 13 部位 = 14 クラスをターゲットに CoaT / ViT 系モデルで学習します。 - モデルバージョンを簡単に整理すると:
- Train v6: ベースラインモデル。OOF 予測から怪しいスライスを集めて
Filter v6(filt1.csv)を作成。 - Train v8 & v9: v6 で絞ったデータ+全体データを使い、より強いモデルを学習。OOF を使って
Filter v8を作成。 - Train v10 & v13: v8 Filter などを利用し、さらにチューニングしたモデル。
- Train v6: ベースラインモデル。OOF 予測から怪しいスライスを集めて
-
これらのうち、特に v8 / v9 / v10 / v13 が try7 のベストモデル群とされます。
- 各モデルで OOF(Out-of-Fold)予測の OUTPUTS / TARGETS / IDS を全て保存しておき、後の pseudo-distillation に用います。
Pseudo‑Distillation(紫)
-
ベストモデル群の OOF 確率を読み込み、モデル間で平均して教師確率ベクトル(teacher_prob)を作成し、pseudo label として保存します。
- この teacher_prob を元ラベルと並べて新しい教師信号とし、知識蒸留(distillation)を行います。対象モデルは:
- MaxViT-tiny v1
- MaxViT-small v1
- CoaT-Lite-Medium v3
- これらのモデルに対し、Segmentation Crop で切り抜いた ROI 画像を入力し、
- 0/1 の hard ラベル(train_sampled / train.csv)
- pseudo teacher の確率分布(teacher_prob)
の両方を予測させます。損失は例えば以下のような形です:
loss = BCE_with_logits(logits, hard_targets) \
+ λ * KL(sigmoid(logits) || teacher_prob)
- こうして try7 のアンサンブルが持っていた知識を、より強力な構造(MaxViT / CoaT v3)を持つ 1 モデルに蒸留することで、try8 の強力な最終モデル群が完成します。
Inference Ensemble(ピンク)
-
テストデータに対しては、まず ViT-Small-Plus DINOv3 Crop Model を走らせて bbox(ROI)を推定し、その bbox でスライスを crop してモデル入力を作成します。
- 次に、以下の 4 系統のモデルから出力を取得します:
- try7 CoaT-Lite-Medium v13(fold 2, 3)
- try8 CoaT-Lite-Medium v3(fold 0, 1)
- try8 MaxViT-small v1(fold 2, 3)
- try8 MaxViT-tiny v1(fold 0, 1)
(図には fold 割り当てが明記されており、合わせて 0〜3 の 4 fold をカバーしています。)
- 各モデルの 14 クラス確率をアンサンブルして、最終的な提出用の予測を作成します。
5 位解法
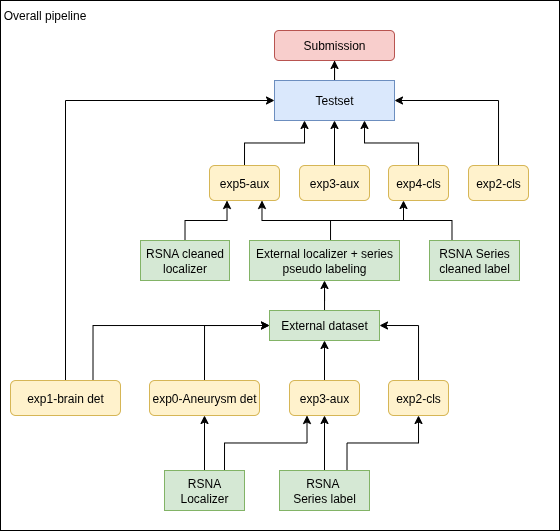
RSNA データから基礎モデルの作成
RSNA Localizer / RSNA Series Label(緑)
- RSNA 公式で提供されているラベル(シリーズレベルラベルおよび動脈瘤中心座標)です。
動脈瘤検出(exp0)
-
Localizer の重心座標の前後 ±10 スライス程度を表示し、LabelImg で動脈瘤を囲む bbox を人手でアノテーションします。
-
得られた bbox を教師データとして、YOLOv11(物体検出モデル)を学習します。
マルチクラス分類 + マルチタスクモデル(exp2 / exp3)
-
exp2-cls: ViT Large / EVA Large による 14 クラス分類モデル。陰性シリーズは「全スライス」を使用し、陽性シリーズは exp0 で作成した bbox を含むスライスのみを使用して学習します。
-
exp3-aux: MIT-B4 + FPN で構成される「分類 + セグメンテーション」のマルチタスクモデルです。分類ラベルは 14 クラス、セグメンテーションラベルは exp0 の bbox を塗りつぶして作成したマスクです。
脳検出モデル(exp1)
-
各シリーズの全スライスを平均して 1 枚の画像を生成し、その画像に対して脳領域の bbox を人手でアノテーションします。
-
その bbox を教師として YOLOv5n で脳検出モデルを学習します。
外部データセットの活用
-
Lausanne_TOFMRA, Royal_Brisbane_TOFMRA の 2 つの外部 TOF-MRA データセットを使用します。最初はラベルがなく、どこに動脈瘤があるかは分かりません。
-
RSNA で学習済みの
- exp2-cls(シリーズレベル 14 クラス分類)
- exp3-aux(分類 + セグメンテーション)
- exp0-Aneurysm det(YOLO 検出)
を使い、外部データセットに pseudo labeling を施します。前 2 つでシリーズレベルの aneurysm 有無を推定し、exp0 で bbox を予測し位置を推定します。
RSNA ラベルのクリーニング
-
pseudo label の発想を RSNA の元トレーニングデータにも適用し、ラベルの誤りを修正します。
-
具体的には、RSNA の Negative シリーズのうち、exp2-cls と exp3-aux の両方で
aneurysm positive > 0.9となるシリーズを探します。これらはラベルミスである可能性が高いとみなし、Negative → Positive に変更し、exp0 の YOLO で bbox を取得してラベルを付与します。
クリーニングした RSNA + 外部 pseudo データで再学習
- クリーニング済み RSNA データセットと、外部 pseudo labeled データセットを合わせて、exp4-cls / exp5-aux を新たに学習します。どちらも exp2-cls / exp3-aux の強化版に相当します。
テストセットでの推論と提出
- Kaggle テストデータに対し、exp4-cls / exp5-aux / exp0 / exp1 など、計 4 つ前後のモデルで予測を出し、それらをアンサンブルして最終的な提出用予測とします。
6 位解法
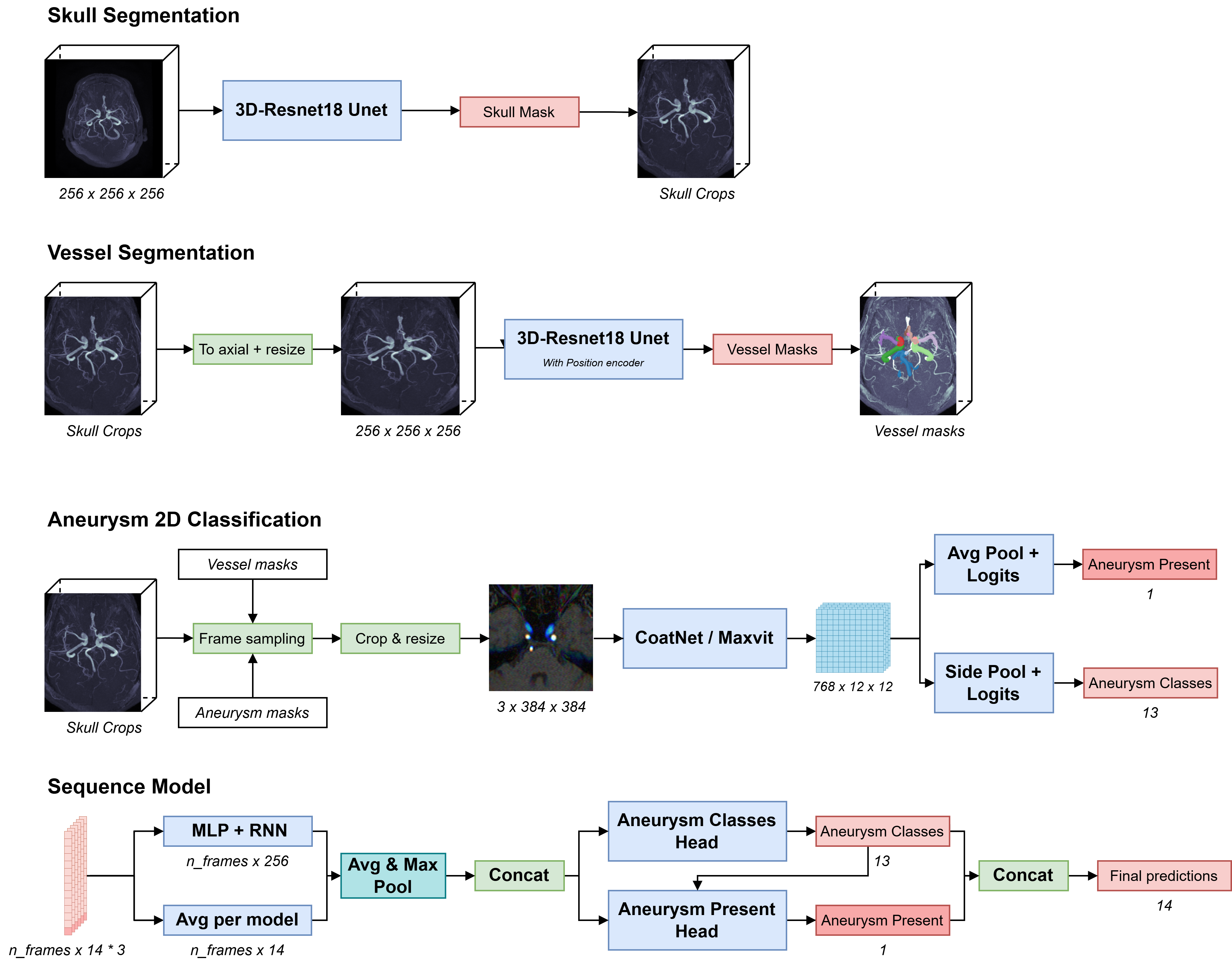
6 位解法はコードが共有されておらず、詳細を深追いしきれなかったので、論文から読み取れた範囲でまとめます。
Skull Segmentation
- volume を 3D-ResNet18 U-Net に入力し、頭蓋骨マスクを推定します。これにより、「頭蓋骨以外の部分」が中心になるように ROI を自動的に切り出せます。
Vessel Segmentation
-
ROI 化した volume を axial 方向に揃えてリサイズし、左右方向の位置エンコーディングを付加したうえで、3D-ResNet18 U-Net に入力し、血管マスクを得ます。
-
得られた血管マスクは、訓練中に「良い Negative(血管はあるが動脈瘤はない場所)」をサンプリングするためにも使用されます。
Aneurysm 2D Classification
-
1 症例の中で動脈瘤が写っているスライスはごく一部で、さらに「画面の端」や「ほとんど何も写っていない」スライスも多く含まれます。そのまま全スライスを使うとクラス不均衡がひどくなるため、陽性フレームを漏らさず拾いつつ、陰性フレームも適度に難しいものだけを選ぶ ことが重要になります。
-
ここでは、複数スライスをまとめたものを 1 つの「フレーム」と呼んでいます(この用語が論文の完全な定義と一致しているかは自信がありません)。
- 陽性フレーム:
- 与えられた動脈瘤位置情報から手動で修正した動脈瘤マスクを使い、そのマスクを含むスライスと、その前後数スライスを
near-positiveとして取得します。
- 与えられた動脈瘤位置情報から手動で修正した動脈瘤マスクを使い、そのマスクを含むスライスと、その前後数スライスを
- 陰性フレーム:
- 血管マスクを含むスライスのうち、ある閾値以上の血管画素を含んでいるものだけを選びます。これにより、「血管はしっかり写っているが動脈瘤はない」ような難しめの陰性サンプルを集められます。
-
こうして作成した陽性・陰性フレームをさらに小さなパッチに固定比率で切り出し、リサイズし、隣接 2 フレームを 3 チャンネル画像にまとめるような前処理を行っています。
- 各 2D フレームを CoAtNet や MaxViT に入力します。これらのネットワークには、相対位置エンコーディング用の MLP が付加されています。ネットワークの最後で特徴マップを 2 つのヘッドに分岐し、それぞれ「動脈瘤の有無」と「部位クラス」を推定しています。
Sequence Model
-
各 3D volume は多数の 2D フレームに分割されているので、フレーム単位の予測を患者単位にまとめるためのシーケンスモデルを用います。
-
まず、各フレームの出力を時系列順に並べ、これを MLP + RNN に入力します。
-
RNN の最終出力と、「単純にフレームごとの出力を平均したもの」を組み合わせ、それを分類ヘッドに入力して最終的な予測を得ます。
7 位解法

7 位解法はパイプライン全体を示す図がなく、ポイントだけ簡単にまとめます。
モデルについて
-
特徴的なのは、3D nnU-Net を使ってヒートマップ回帰を行っている 点です。
-
nnU-Net はもともとセマンティックセグメンテーション用で、整数ラベル(クラス ID)を前提とします。
-
そこでクラスごとに 0/1 マスクを作成し、それに対してユークリッド距離変換をかけて「距離マップ」を生成し、あらかじめ決めた半径の範囲を blob 化することで「連続値のピーク(ヒートマップ)」のようなラベルを作っています。
ROI について
- 他の解法と比べると、ROI 抽出はかなりシンプルです。具体的には:
- 画像の物理座標の中心を求め、
- その位置から上方向(頭頂側)に一定量だけずらした位置を中心として、
[200, 160, 160] mmの立方体を単純にくり抜く、
というロジックになっています。
まとめ
- ほぼ全ての上位解法で、ROI 抽出 → 本タスク(分類 or セグメント + 集約)という マルチステージ構成 が採用されていました。
- 1st stage では、nnU-Net や U-Net 系、YOLO、2D/3D ResNet などを駆使して「頭蓋骨や血管・脳」をきれいに切り出し、そこから動脈瘤がありそうな領域をさらに絞り込んでいます。
- 2nd stage では、3D nnU-Net backbone + attention、3D ResNet、高性能 2D ViT/MaxViT 系などを利用し、さらに pseudo label・distillation・外部データを組み合わせてモデルを強化していました。
参考資料・リンク
※ 記事中の各解法の詳細は、必ず元の Kaggle Discussion / 論文を参照してください(本記事は図を中心にした私的な要約です)。もし明らかな誤りや「ここはこう解釈したほうが近いかも」といった点があれば、教えていただけると嬉しいです。